Documenti di Didattica
Documenti di Professioni
Documenti di Cultura
Trasporto A One e Degradazione Delle Proteine
Caricato da
Marianna MannaTitolo originale
Copyright
Formati disponibili
Condividi questo documento
Condividi o incorpora il documento
Hai trovato utile questo documento?
Questo contenuto è inappropriato?
Segnala questo documentoCopyright:
Formati disponibili
Trasporto A One e Degradazione Delle Proteine
Caricato da
Marianna MannaCopyright:
Formati disponibili
Pag.
1098 a 1108
La cellula eucariotica possiede numerosi compartimenti ognuno dotato di specifici gruppi distinti di enzimi e proteine che, ad eccezione di quelli prodotti nei mitocondri e nei plasmidi, vengono sintetizzati nel citosol dai ribosomi e vengono solo successivamente indirizzate al compartimento a cui apparterranno. Le proteine destinate alla secrezione, allintegrazione nella membrana plasmatica o allinclusione nei lisosomi hanno in comune i primi passaggi di una via enzimatica che comincia nel ER; le proteine destinate ai mitocondri, ai cloroplasti o al nucleo utilizzano 3 meccanismi separati mentre le proteine destinate al citosol semplicemente restano dove vengono sintetizzate. Il trasporto a destinazione viene mediato da una breve sequenza amminoacidica detta sequenza segnale che dirige la proteina verso la sua appropriata destinazione nella cellula e viene rimossa o durante il trasporto oppure dopo che la proteina ha raggiunto la sua destinazione finale. Se le proteine devono essere trasportate ai mitocondri, ai cloroplasti o nel ER la sequenza segnale localizzata allestremit ammino-terminale del polipeptide neosintetizzato. La destinazione cos come la degradazione delle proteine determinata da segnali molecolari presenti nella struttura di ciascuna proteina; infatti considerando due diverse proteine se invertiamo le sequenze segnale per il trasporto osserveremo che le proteine si dirigeranno esattamente dove indica la loro sequenza segnale, a prescindere dalla natura delle proteine stesse. Il trasporta a destinazione di molte proteine come la maggior parte delle proteine lisosomiali, di membrana o destinate alla secrezione, ha inizio nel ER. Queste possiedono una sequenza segnale ammino-terminale che le contraddistingue e che permette loro di essere traslocate nel lume dellER: tale sequenza ha unestremit carbossi-terminale contrassegnata da un sito di scissione che il punto in cui lazione di una proteasi specifica pu rimuovere tale sequenza in seguito alla traslocazione. Le sequenze segnale variano in lunghezza dai 13 ai 36 residui aa ma hanno in comune 3 caratteristiche:
1) Una sequenza di aa idrofobici lunga 10-15 residui; 2) Uno o pi aa carichi positivamente posti generalmente vicino allestremit amminica che precede la sequenza idrofobica; 3) Una breve sequenza, relativamente polare, che posta vicino al sito di scissione e possiede aa aventi corte catene laterali come Ala nelle posizioni
pi vicine al sito di scissione.
Tutte le proteine che contengono sequenze di questo tipo vengono indirizzate al ER.
Meccanismo di indirizzo :
La sintesi proteica inizia per tutte le proteine sui ribosomi liberi; nella fase precoce del processo per se compare la sequenze segnale per lindirizzamento nel ER, che si trova allestremit ammino-terminale del peptide in sintesi, il ribosoma e la sequenza vengono legati da un voluminoso complesso chiamato particella di riconoscimento del segnale o SRP (signal recognition particle) che un complesso a forma di bastoncino che contiene un RNA di 300 nucleotidi e 6 proteine diverse: una subunit proteica di questo complesso si lega direttamente alla sequenza segnale e ne inibisce lallungamento bloccando stericamente lentrata degli amminoacil-tRNA ed inibendo la peptidil transferasi; unaltra subunit invece si lega al GTP ed in seguito lo idrolizza. Il legame della SRP al GTP determina quindi la sospensione temporanea dellallungamento quando il peptide lungo circa 70 aa e la sequenza segnale completamente emersa dal ribosoma e dirige il ribosoma, ancora legato allmRNA, ed il polipeptide incompleto verso un gruppo di recettori specifici per la SRP con legato il GTP sulla superficie citosolica dellER; questo recettore un eterodimero composto da una subunit ed una subunit che legano ed idrolizzano le molecole di GTP utili per questo processo. Il polipeptide passa quindi al complesso di traslocazione del peptide nellER che interagisce direttamente con il ribosoma per cui la SRP, che non ha pi unutilit, si dissocia in seguito allidrolisi di GTP da parte sia della SRP che del recettore di SRP. Con un processo che utilizza lenergia dellATP (idrolizzato dal recettore per la SRP) procede lallungamento del peptide, che grazie al complesso di traslocazione viene introdotto direttamente allinterno del lume dellER, fino alla completa sintesi della proteina. La
sequenza segnale viene successivamente rimossa da una peptidasi specifica presente allinterno del lume dellER ed il ribosoma dissociato pu essere riciclato. Le proteine cos entrate nellER subiscono ulteriori modificazioni : in seguito alla rimozione della sequenza segnale i polipeptidi vengono avvolti con la formazione di legami disolfuro e molte proteine vengono glicosilate. La glicosilazione consiste nellaggiunta a tali polipeptidi di oligosaccaridi legati a residui di Asn che prendono poi il nome di glicoproteine; gli oligosaccaridi legati in N sono molto diversi tra loro ma le vie per la loro formazione hanno il primo passaggio in comune :
Il processo porta alla formazione di un nucleo oligosaccaridico, contenente 14 residui (2 di N-acetilglucosammina, 3 di glucosio e 9 di mannosio), legato ad una molecola di dolicolo fosfato ; i precursori della sintesi, che forniscono residui di mannosio e glucosio alloligosaccaride in via di formazione, sono monosaccaridi uniti a molecole di dolicolo fosfato allinterno del lume dellER e legati a nucleotidi difosfato allinizio del processo, nella parte citosolica. Le prime fasi di aggiunta di oligosaccaridi avvengono infatti a livello della superficie citosolica dellER ad opera di 2 UDP-N-acetilglucosammina e 5 GDPmannosio che forniscono 2 molecole di N-acetilglucosammina e 5 molecole di mannosio rispettivamente fino a che loligosaccaride, ancora incompleto, viene traslocato allinterno della membrana e qui si ha il completamento del nucleo oligosaccaridico ovvero dove molecole di dolicolo fosfato permettono laggiunta di 4 molecole di mannosio e 3 molecole di glucosio. Loligosaccaride completo, legato alla molecola di dolicolo fosfato, deve adesso essere trasferito alla proteina e ci avviene ad opera di una transferasi che localizzata sulla superficie dellER (che per questo non pu catalizzare la glicosilazione delle proteine citoplasmatiche); quindi loligosaccaride dalla molecola donatrice, il dolicolo fosfato, viene legato ad un residuo di Asn della proteina allinterno dellER attraverso un legame N-glicosidico. Cinque residui saccaridici (quelli cerchiati in rosso) rimangono immodificati e sono quindi conservati nella struttura finale di tutti gli oligosaccaridi uniti con legami N-glicosidici; gli altri invece subiscono ulteriori modificazioni sia nellER che nel complesso di Golgi. Il dolicolo pirofosfato che viene rilasciato prima traslocato nella faccia citosolica della membrana dove un fosfato viene rimosso idroliticamente e viene cos rigenerato il dolicolo fosfato pronto ad unaltra glicosilazione. LO-glicosilazione delle proteine invece avviene principalmente nel complesso di Golgi nel citosol; infatti solo poche vengono O-glicosilate nellER. Diversi antibiotici interferiscono con il processo di glicosilazione; uno tra questi la tunicamicina che mima la struttura dellUDP-N-acetilglucosammina e blocca quindi la prima fase del processo. Una volta modificate nellER le proteine giungono, trasportate da vescicole, al lato cis del complesso di Golgi; qui vengono aggiunti gli oligosaccaridi legati in O e modificati quelli legati in N. Ed proprio nel Golgi che le proteine vengono smistate ed indirizzate alle rispettive destinazioni finali e ci viene compiuto attraverso la distinzione delle caratteristiche strutturali differenti della sequenza segnale delle proteine precedentemente rimossa nellER. Il processo di segregazione stato meglio definito nel caso delle idrolasi, proteine destinate a raggiungere i lisosomi; lidrolasi, che una glicoproteina, dopo esser giunta e modificata nel Golgi viene riconosciuta, grazie ad alcune caratteristiche non ancora definite della sua struttura tridimensionale, da una fosfotransferasi che fosforila alcuni residui di mannosio presenti negli oligosaccaridi legati in N di questo enzima : la presenza di uno o pi residui di mannosio 6-fosfato dirige questi enzimi ai lisosomi. Infatti a livello della membrana del Golgi presente un recettore che riconosce il segnale del mannosio 6-fosfato e lega lidrolasi; le vescicole cos contenenti i complessi recettore-idrolasi segregano verso il versante trans del Golgi e vengono rilasciate come vescicole di secrezione. Allinterno di ogni vescicola il pH molto basso e la rimozione, ad opera di una fosfatasi, dei gruppi fosforici legati ai residui di mannosio 6-fosfato, determinano la dissociazione del complesso recettore-idrolasi; il recettore torna al complesso di Golgi e le vescicole si dirigono ai lisosomi e qui rilasciano le idrolasi. Loligosaccaride legato in N determinante in questa via di trasporto per la destinazione delle vescicole; infatti in cellule trattate con tunicamicina, le vescicole non raggiungono i lisosomi ma vengono secrete. Le sequenze segnale ammino-terminali svolgono un ruolo chiave anche per il trasporto di proteine destinate ai mitocondri ed ai cloroplasti; infatti, sebbene tali organelli contengano DNA, la maggior parte delle loro proteine vengono codificate dal DNA nucleare e quindi sintetizzate nel citosol della cellula che ospita tali organelli. Queste proteine, a differenza della proteine destinate allER, vengono completamente sintetizzate e rilasciate dai ribosomi prima di essere trasportate; solo successivamente, dopo che la sequenza segnale stata rimossa, vengono legate da proteine chaperone del citosol e rilasciate a livello di recettori situati sulla superficie esterna dellorganello a cui sono destinate. La comunicazione molecolare tra nucleo e citoplasma in una cellula eucaristica molto intensa : la molecole di RNA vengono sintetizzate nel nucleo e poi esportate nel citoplasma; le proteine ribosomiali sintetizzate dai ribosomi nel citosol vengono portate nel nucleo e nel nucleolo sono assemblate ad rRNA per costituire le subunit 60S e 40S che sono poi trasportate nel citosol; molte proteine nucleari come RNA e DNA polimerasi, istoni, topo isomerasi, proteine che regolano lespressione genica, vengono sintetizzate nel citosol e poi trasportate nel nucleo. Negli eucarioti ogni divisione cellulare porta alla rottura ed alla ristrutturazione dellinvolucro nucleare e di conseguenza alla dispersione delle proteine contenute nel nucleo. Per garantire che in seguito alla ristrutturazione dellinvolucro tutte le proteine nucleari disperse possano rientrare nel nucleo, la sequenza segnale che indirizza le proteine verso il nucleo, detta sequenza di localizzazione nucleare o NLS, non viene rimossa in seguito al raggiungimento della proteina a destinazione. Tale sequenza infatti non localizzata come le altre sequenze segnale allestremit ammino-terminale della proteina ma pu trovarsi in qualsiasi punto della sequenza primaria della proteina e, sebbene possa variare considerevolmente, in generale risulta essere costituita da 4 a 8 residui amminoacidici che includono alcuni residui basici consecutivi (Arg o Lys).
Meccanismo di trasporto :
Un certo numero di proteine vanno avanti ed indietro tra citosol e nucleo e permettono il trasporto. Una proteina con un appropriato segnale di localizzazione nucleare (NLS) viene legata ad un complesso, limportina. Questa proteina nella sua forma eterodimerica costituita dalle subunit e e funziona come un recettore solubile per il trasporto delle proteine verso il nucleo; la subunit si lega alle proteine che portano lNLS nel citosol ed alla subunit e tale complesso si lega ad un poro nucleare e viene traslocato attraverso il poro, tramite un meccanismo energia-dipendente mediato dalla GTPasi Ran. Durante la traslocazione limportina si dissocia nelle subunit che la costituiscono e mentre la proteina trasportata resta nel nucleo le subunit e dellimportina vengono esportate dal nucleo per ripetere il processo.
I batteri possono anchessi indirizzare alcune loro proteine sia alle membrane (interna ed esterna), sia allo spazio periplasmico compreso tra queste membrane, sia al mezzo extracellulare. Per effettuare questo trasporto essi utilizzano sequenze segnale allestremit ammino-terminale delle proteine sintetizzate, proprio come gli eucarioti. Sebbene alcune proteine seguano una via che utilizza componenti di riconoscimento del segnale e recettori omologhi a SRP ed al suo recettore negli eucarioti; la maggior parte delle proteine esportate segue questo modello: Una proteina che deve essere esportata durante la sintesi si ripiega molto lentamente in quanto la sua sequenza ammino-terminale ne impedisce lavvolgimento. Questo permette il legame della proteina chaperone solubile SecB che si lega alla sequenza segnale di questa proteina o ad altre caratteristiche della sua struttura ripiegata in modo incompleto. Una volta raggiunta la membrana SecB rilascia la proteina a SecA, una proteina associata alla superficie interna della membrana citoplasmatica che agisce sia da ATPasi di traslocazione che da
recettore (lATPasi si trova sulla faccia citoplasmatica in quanto qui presente
lATP). Quando dissociata da SecB ma legata a SecA la proteina viene inserita nella membrana allinterno di un complesso di traslocazione, costituito dalle proteine Sec Y,E e G, per un segmento di circa 20 aa e gradualmente viene spinta allinterno della membrana a tratti di 20 aa, meccanismo mediato da SecA che lega ATP e lo idrolizza ed questo che fornisce lenergia, insieme al potenziale elettrochimico transmembrana, per la traslocazione della proteina. Lidrolisi di ATP da parte di SecA fornisce lenergia per una modificazione conformazionale che provoca il ritiro di SecA dalla membrana ed il rilascio del polipeptide. Le proteine possono anche essere importate nella cellula dal mezzo extracellulare secondo un processo detto endocitosi. Alcune proteine come le LDL (lipoproteine a bassa densit), la transferrina (proteina che trasporta il ferro), gli ormoni peptidici, le proteine circolanti destinate alla degradazione si legano a recettori situati su invaginazioni sulla superficie della membrana dette invaginazioni rivestite (coated pits) che contengono appunto preferenzialmente recettori endocitici piuttosto che altre proteine di superficie. Queste invaginazioni sul lato citosolico sono rivestite da un reticolo formato da clatrina, una proteina le cui unit sono i triskelion (una struttura a tre gambe costituita da 3 catene leggere e 3 catene pesanti) che si associano a formare un reticolo poliedrico chiuso.
Le proteine iniziano ad occupare quindi i recettori facendo aumentare di dimensioni linvaginazione e di conseguenza anche il reticolo di clatrina fino a che una vescicola endocitica si stacca dalla membrana e si dirige nel citoplasma; la clatrina viene rimossa da enzimi specifici e le vescicole si fondono con gli endosomi dove il basso pH (mantenuto tale dallattivit delle ATPasi) facilita la dissociazione dei recettori dalle loro proteine bersaglio che procedono poi per vie separate a seconda del sistema considerato. La transferrina ed il suo recettore vengono riciclati; alcuni ormoni, fattori di crescita ed immunocomplessi vengono degradati insieme con i loro recettori dopo aver svolto le loro funzioni; le LDL vengono degradate dopo che il colesterolo ad esse associato viene rilasciato nel sito di destinazione mentre il loro recettore viene riciclato.
Per impedire laccumulo di proteine anormali o non necessarie e facilitare il riciclaggio degli amminoacidi, le proteine in una cellula vengono continuamente
degradate. La maggior parte viene riciclata rapidamente in rapporto allemivita di una cellula eucariotica (che varia da mezzo minuto a molti giorni) ma
alcune particolarmente stabili possono durare per tutta la vita della cellula (ad esempio lemoglobina dura per 110 giorni, che lemivita di un eritrocita). Quelle degradate pi rapidamente sono le proteine che presentano anomalie, come linserzione di aa sbagliati durante la sintesi, o che hanno subito danni durante il loro funzionamento o gli enzimi che agiscono da regolatori in una via metabolica. La degradazione avviene ad opera di sistemi citosolici ATP-dipendenti e nei vertebrati esiste anche un secondo sistema che opera nei lisosomi , non specifico e serve a riciclare gli aa delle proteine di membrana, di quelle extracellulari e quelle con emivita particolarmente lunga. In E. coli molte proteine vengono degradate da una proteasi ATP-dipendente detta Lon che viene attivata in presenza di proteine difettose o con breve emivita (ricambio rapido); ogni legame peptidico viene scisso grazie allidrolisi di 2 molecole di ATP (non ancora chiara la funzione dellidrolisi di ATP). Una volta che la proteina viene ridotta a brevi peptidi inattivi altre proteasi ATP-dipendenti completano la degradazione. Negli eucarioti la via ATP-dipendente, molto regolata e specifica a differenza di quella operante nei lisosomi, coinvolge la proteina ubiquitina che una delle proteine note pi conservate durante levoluzione, infatti essenzialmente identica in organismi molto diversi come i lieviti e luomo. Attraverso una via ATP-dipendente che utilizza 3 enzimi distinti lubiquitina viene legata covalentemente alla proteina da degradare :
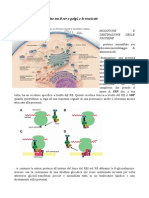
1 Avviene dapprima l' attivazione dell'ubiquitina con consumo di ATP. In questa prima tappa implicato un enzima 1 (E1) che si lega all'ubiquitina
tramite un legame tioestere* ad alta energia. il carbossile C-terminale di un residuo di Gly dell'ubiquitina che forma un legame tioestere con una cisteina (Cys) di E1. L'ATP viene scisso in AMP + PPi e la pirofosfatasi inorganica idrolizzer ancora il PPi, per cui in totale vengono scissi due legami pirofosforici dellATP. L'E1 viene denominato enzima di attivazione dell'ubiquitina. Questo processo non sembra avere un ruolo regolativo e gli organismi eucariotici hanno solo poche isoforme E1.
Nella seconda tappa interviene l enzima 2 (E2), definito enzima di coniugazione dell'ubiquitina, che agisce in coniugazione con la proteina accessoria E3 formando un complesso detto ubiquitina ligasi, viene sostituito ad E1 in una reazione che prevede la formazione di un legame tioestere ad alta energia tra una Cys di E2 e il gruppo carbossilico del residuo di Gly dellubiquitina legato ad E1. La Cys che forma il legame la numero 88; ciascuna specie contiene numerosi enzimi di tipo 2, che presentano elevata omologia di sequenza solo nella regione che circonda la Cys88, invece nella parte restante della proteina in pratica non riscontrabile omologia di sequenza. Quindi tale zona che circonda la Cys-88 deputata al riconoscimento dellubiquitina. In definitiva questa tappa-2 porta al trasferimento dellubiquitina da E1 a E2.
Nel complesso ubiquitina ligasi (E2-E3), la componente E3 lega specifici segnali di degradazione nelle proteine substrato ed aiuta E2 a legare l'ammino gruppo di qualche Lys sulla proteina che deve essere degradata mediante un legame ammidico (isopeptidico, vedi cerchio rosso).Si forma cos una catena multiubiquitina dove ciascun residuo C-terminale di ubiquitina legato ad una Lys specifica della molecola di ubiquitina che la precede, producendo una serie lineare di coniugati ubiquitina-ubiquitina. Vi sono circa 30 enzimi distinti E2 che si associano con centinaia di proteine E3 diverse; ogni complesso riconosce segnali di degradazione differenti ed alcuni enzimi sono anche localizzati unicamente in specifici compartimenti cellulari, di cui riflettono le funzioni specializzate. *formazione legame tioestere:
Una volta ubiquitinate le proteine vengono degradate da un grande complesso detto proteasoma 26S, una proteasi molto abbondante dipendente da ATP presente in molte copie disperse nel citosol e nel nucleo e che ha come bersaglio anche le proteine dell ER ovvero quelle proteine che non riescono a ripiegarsi o ad assemblarsi in modo appropriato. Un proteasoma costituito da un cilindro cavo centrale che il nucleo del proteasoma 20 S formato da
subunit proteiche e che si assemblano come una pila cilindrica di 4 anelli eptamerici (gli anelli esterni sono costituiti da 7 subunit mentre quelli
interni da 7 subunit e 3 delle 7 subunit possiedono attivit proteasica ciascuna con diverse specificit di substrato). Ciascuna estremit di tale cilindro normalmente associata ad un grande complesso proteico, il cappuccio 19 S che contiene 18 subunit, alcune delle quali hanno la funzione di riconoscere e legarsi alle proteine ubiquitinate. Sei di queste subunit, poste vicino al bordo del cilindro, sono ATPasi che si pensa disavvolgano le proteine ubiquitinate e le traslochino nel nucleo per la degradazione. Una caratteristica interessante di questa proteasi la processivit; infatti il proteasoma quando lega il substrato, la proteina ubiquitinata, non si dissocia nelle subunit che lo costituiscono fino a che lintero polipeptide stato tutto convertito in corti peptidi. I cappucci 19 S agiscono da cancelli regolati allentrata della camera proteolitica interna e sono responsabili dellattacco della proteina substrato al proteasoma. I segnali che innescano lubiquitinazione non sono completamene conosciuti ma per molte proteine lidentit del primo residuo ammino-terminale (ovvero quello che rimane dopo la rimozione del residuo di Met e dopo tutte le modificazioni proteolitiche post-traduzionali allestremit ammino-terminale) ha una profonda influenza sullemivita della proteina; ad esempio residui di Met, Gly, Ala, Ser,Thr, Val risultano essere stabilizzanti, ovvero aumentano notevolmente lemivita della proteina che li contiene (>20 ore); a differenza di altri residui come Arg (emivita circa 2 minuti) o Leu, Phe, Asp, Lys (emivita circa 3 minuti) o Pro (emivita circa 7 minuti) oTyr, Glu (emivita circa 10 minuti) o Ile, Gln (emivita circa 30 minuti) che sono fortemente destabilizzanti. Esistono comunque segnali ben pi complessi per lubiquitinazione come la sequenza di distruzione presente nelle cicline che viene riconosciuta dalla DBRP. Risulta quindi chiaro che difetti nel processo di ubiquitinazione potrebbero essere coinvolti in unampia gamma di malattie : unincapacit nel degradare certe proteine che attivano la divisione cellulare (i prodotti degli oncogeni) o una degradazione troppo rapida di proteine che agiscono da soppressori tumorali pu portare infatti alla formazione di tumori.
Potrebbero piacerti anche
- Smistamento Proteine 1Documento11 pagineSmistamento Proteine 1Chiara FerranteNessuna valutazione finora
- 12.4 Ruoli Del RE e Dell'appato Del Golgi Nel Traffico Delle ProteineDocumento2 pagine12.4 Ruoli Del RE e Dell'appato Del Golgi Nel Traffico Delle ProteineGianmarco MengoliNessuna valutazione finora
- 12.1 Il Reticolo EndoplasmaticoDocumento2 pagine12.1 Il Reticolo EndoplasmaticoGianmarco MengoliNessuna valutazione finora
- Biologia GeneraleDocumento4 pagineBiologia GeneraleTereresa2Nessuna valutazione finora
- Trascrizione Del DNA Nei ProcariotiDocumento4 pagineTrascrizione Del DNA Nei ProcariotiBarbaraNessuna valutazione finora
- Reticolo Endoplasmatico, Apparato Del Golgi, LisosomiDocumento11 pagineReticolo Endoplasmatico, Apparato Del Golgi, LisosomiAlessandro LuminelloNessuna valutazione finora
- Trascrizione Del DnaDocumento3 pagineTrascrizione Del DnaClaudia BertenniNessuna valutazione finora
- ReR, Rel, Golgi e VescicoleDocumento5 pagineReR, Rel, Golgi e VescicoleMarco BiscosiNessuna valutazione finora
- Il Re e Il Golgi Nel Traffico Di ProteineDocumento1 paginaIl Re e Il Golgi Nel Traffico Di ProteineCosso GiuliaNessuna valutazione finora
- BiosegnalazioneDocumento12 pagineBiosegnalazioneiotu2547Nessuna valutazione finora
- Bio Chi MicaDocumento59 pagineBio Chi Micafocaxo5386Nessuna valutazione finora
- Ribosomi E La Sintesi Delle Proteine I RibosomiDocumento6 pagineRibosomi E La Sintesi Delle Proteine I RibosomiRosi De LucaNessuna valutazione finora
- Lezione 15Documento5 pagineLezione 15bertasierramaquedaNessuna valutazione finora
- Trascrizione DNA Traduzione RNA MutazioniDocumento7 pagineTrascrizione DNA Traduzione RNA MutazioniGianmarco VindigniNessuna valutazione finora
- Trascrizione e TraduzioneDocumento3 pagineTrascrizione e TraduzioneEMANUELE LA MARCANessuna valutazione finora
- Lezione 11.11Documento16 pagineLezione 11.11ElisaNessuna valutazione finora
- Copia Di ChimicaDocumento18 pagineCopia Di ChimicaViola TalaNessuna valutazione finora
- Costruire Ponti, Non MuriDocumento5 pagineCostruire Ponti, Non MuriAlessia PetragalloNessuna valutazione finora
- 11.1 Apparato Del GolgiDocumento4 pagine11.1 Apparato Del GolgiannaNessuna valutazione finora
- Sistesis de LipidosDocumento2 pagineSistesis de LipidosbertasierramaquedaNessuna valutazione finora
- Capitolo 5 Citologia Dalle DonneDocumento3 pagineCapitolo 5 Citologia Dalle DonneEugenio Stracuzza100% (1)
- Sintesi Lezioni BiochimicaDocumento156 pagineSintesi Lezioni BiochimicaUmberto Sueri100% (1)
- Cap. 7 Membrana PlasmaticaDocumento6 pagineCap. 7 Membrana PlasmaticaRosemarie ScaliNessuna valutazione finora
- App Biol GenDocumento67 pagineApp Biol GenCosimo SavoiaNessuna valutazione finora
- Bersagli CellulariDocumento3 pagineBersagli CellulariDora CandiaNessuna valutazione finora
- Trasporti Di MembranaDocumento10 pagineTrasporti Di MembranaFrancesco BorealeNessuna valutazione finora
- 03 Ribosomi, RE e GolgiDocumento2 pagine03 Ribosomi, RE e GolgiElton Tony KazaziNessuna valutazione finora
- Biologia MolecolareDocumento8 pagineBiologia MolecolareMaria MoiseiNessuna valutazione finora
- Effetti della trombopoietina sul microcircolo coronaricoDa EverandEffetti della trombopoietina sul microcircolo coronaricoNessuna valutazione finora
- Appunti Su Dna e RnaDocumento3 pagineAppunti Su Dna e RnaLuca LeggieroNessuna valutazione finora
- CITOLOGIA E ISTOLOGIA - Lezione 12Documento4 pagineCITOLOGIA E ISTOLOGIA - Lezione 12Federico TosiniNessuna valutazione finora
- BiologiaDocumento6 pagineBiologiaDenise TestaniNessuna valutazione finora
- Appunti MetabolismoDocumento19 pagineAppunti MetabolismoLuca CanegratiNessuna valutazione finora
- Trascrizione Del DnaDocumento3 pagineTrascrizione Del Dnasara nolliNessuna valutazione finora
- Vescicole EndolisosomialiDocumento3 pagineVescicole EndolisosomialiFrancesco BorealeNessuna valutazione finora
- 14.aparato de GolgiDocumento5 pagine14.aparato de GolgibertasierramaquedaNessuna valutazione finora
- Respirazione e Fermentazione-MicrobiologiaDocumento18 pagineRespirazione e Fermentazione-MicrobiologiaSamuel Di sottoNessuna valutazione finora
- Apparato Golgi + LisosomiDocumento24 pagineApparato Golgi + LisosomibracciodiferoNessuna valutazione finora
- La Replicazione Del DNADocumento4 pagineLa Replicazione Del DNAzio peraNessuna valutazione finora
- Risposte A Domande BiochimicaDocumento7 pagineRisposte A Domande BiochimicaLuigia CentolaNessuna valutazione finora
- La Fosforilazione Ossidativa e La FermentazioneDocumento2 pagineLa Fosforilazione Ossidativa e La FermentazioneCristian ChirNessuna valutazione finora
- BiologiaDocumento3 pagineBiologiaFla PessaNessuna valutazione finora
- Sintesi Proteica 2Documento1 paginaSintesi Proteica 2Fabio Picatrix DelaVirgenNessuna valutazione finora
- Acidi NucleiciDocumento6 pagineAcidi NucleiciAllegra BiancoNessuna valutazione finora
- Genetica Modulo 2 PDFDocumento4 pagineGenetica Modulo 2 PDFGiorgioNessuna valutazione finora
- Il Metabolismo Del GlucosioDocumento3 pagineIl Metabolismo Del GlucosiojolandavigoritaNessuna valutazione finora
- Dal DNA Alla Genetica Dei MicrorganismiDocumento5 pagineDal DNA Alla Genetica Dei MicrorganismiMarino MariniNessuna valutazione finora
- Miv Sistemi Di SecrezioneDocumento11 pagineMiv Sistemi Di Secrezioneantonio nennaNessuna valutazione finora
- 12Documento2 pagine12benedettamari00Nessuna valutazione finora
- Metabolismo CellulareDocumento5 pagineMetabolismo CellulareLeonardo TovoNessuna valutazione finora
- OsmolaritàDocumento7 pagineOsmolaritàCarlotta GhibelliniNessuna valutazione finora
- BatteriologiaDocumento98 pagineBatteriologiaFilippoNessuna valutazione finora
- Reticolo EndoplasmaticoDocumento5 pagineReticolo EndoplasmaticoEdoardo MalaguerraNessuna valutazione finora
- Traduzione ConclusioneDocumento4 pagineTraduzione ConclusioneGina D'antuonoNessuna valutazione finora
- Trasduzione Del SegnaleDocumento4 pagineTrasduzione Del Segnalesimone_greco_10Nessuna valutazione finora
- Dna e Sintesi ProteicaDocumento6 pagineDna e Sintesi ProteicaLuigi BorgeseNessuna valutazione finora
- Microbiologia GeneraleDocumento49 pagineMicrobiologia GeneraleAntonio Chimenti100% (1)
- UntitledDocumento115 pagineUntitledilaria zegaNessuna valutazione finora
- Domande MicrobiologiaDocumento43 pagineDomande MicrobiologiaAnonymous EsRgB8Nessuna valutazione finora
- Carboidrati, Proteine e PolisaccaridiDocumento8 pagineCarboidrati, Proteine e PolisaccaridiFederica MellaceNessuna valutazione finora
- PragliaDocumento162 paginePragliadanimuraNessuna valutazione finora
- Appunti Di Italiano ZibaldoneDocumento2 pagineAppunti Di Italiano ZibaldoneMirea DestratisNessuna valutazione finora
- Scheda 09 GrammaticaDocumento2 pagineScheda 09 GrammaticaMatías Costantini ReschiaNessuna valutazione finora
- Docimologia CompletoDocumento56 pagineDocimologia CompletoSybille ZeniNessuna valutazione finora
- Il Libro Dei Proverbi e Detti Antichi Napoletani - Parte 1 - DUEDocumento22 pagineIl Libro Dei Proverbi e Detti Antichi Napoletani - Parte 1 - DUEantonioNessuna valutazione finora
- Il FascismoDocumento4 pagineIl Fascismofederico romanoNessuna valutazione finora
- Manuale Osservazione VariabiliDocumento83 pagineManuale Osservazione Variabilipolpoldino100% (1)
- Elenco Piante Antiche Ago 2011Documento42 pagineElenco Piante Antiche Ago 2011jonkjNessuna valutazione finora