Documenti di Didattica
Documenti di Professioni
Documenti di Cultura
DNA
Caricato da
Li Clemente0 valutazioniIl 0% ha trovato utile questo documento (0 voti)
77 visualizzazioni2 pagineTitolo originale
DNA.docx
Copyright
© © All Rights Reserved
Formati disponibili
DOCX, PDF, TXT o leggi online da Scribd
Condividi questo documento
Condividi o incorpora il documento
Hai trovato utile questo documento?
Questo contenuto è inappropriato?
Segnala questo documentoCopyright:
© All Rights Reserved
Formati disponibili
Scarica in formato DOCX, PDF, TXT o leggi online su Scribd
0 valutazioniIl 0% ha trovato utile questo documento (0 voti)
77 visualizzazioni2 pagineDNA
Caricato da
Li ClementeCopyright:
© All Rights Reserved
Formati disponibili
Scarica in formato DOCX, PDF, TXT o leggi online su Scribd
Sei sulla pagina 1di 2
AISLAMIENTO DE DNA DE ORIGEN VEGETAL Y SU CARACTERIZACIÓN ESPECTROFOTOMÉTRICA
IDENTIFICACIÓN DE BASES PÚRICAS Y PIRIMÍDICAS POR CROMATOGRAFÍA EN CAPA FINA
Objetivo: etilendiaminotetraacético, que es un agente
Aislar, purificar y caracterizar quelante, que puede crear complejos con iones
espectrofotométricamente DNA de guayaba divalentes, dado que el Ca2+ es un cofactor
Identificar por cromatografía en capa fina las requerido por las nucleasas, su secuestro por el
bases nitrogenadas del DNA y RNA EDTA evita la degradación del DNA, el NaCl …
aprovechando su propiedad para absorber El almidón de la guayaba se hidroliza por la
luz UV. acción de la enzima amilasa, la cual rompe los
Introducción: enlaces α 1, 4.
Los ácidos nucleicos representan la cuarta gran La solución saturada de NaCl provoca la
clase de macromoléculas. Éstas, igual que las precipitación salina de proteínas y el SDS al 10
proteínas y los polisacáridos, contienen % es un compuesto tensioactivo aniónico
múltiples unidades monoméricas similares que antipático, toma la parte lipídica de la célula y
se unen en forma covalente para producir forma semimicelas, además rompe enlaces no
polímeros grandes. El DNA y RNA son muy covalentes de las proteínas, por lo que deshace
apropiados para funcionar como portadores de la membrana y libera el contenido de la célula.
información genética en virtud de sus Despues de la centrifugación se obtienen tres
estructuras covalentes. Los nucleótidos tienen fases: En la superior se encuentra el DNA, en la
tres componentes: un azúcar con cinco media las proteínas coaguladas y en la inferior
carbonos, uno o más grupos fosfato y un la mezcla de cloroformo: alcohol isoamílico.
compuesto nitrogenado débilmente básico El sobrenadante se vierte en alcohol 96° para la
llamado base, las cuales son derivadas de precipitación de los polímeros biológicos entre
pirimidina (tiene un solo anillo de cuatro átomos los cuales se encuentra el DNA de guayaba
de carbono y dos de nitrógeno) y purina (tiene
un sistema de anillos fundidos de pirimidina y de Resultados:
imidazol). Los dos tipos de bases son no Se anexa:
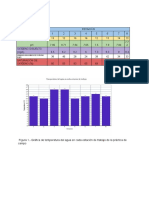
saturados, con dobles enlaces conjugados. Esta Grafica 1. Espectro de absorción de DNA de
propiedad hace que los anillos sean planos, y guayaba.
también explica su capacidad de absorber la luz
ultravioleta.
El RNA está formado por: el azúcar D-Ribosa,
bases nitrogenadas (adenina, guanina, citosina
o uracilo) y el grupo fosfato.
El DNA esta formado por: el azúcar
desoxirribosa (el prefijo desoxi indica que el
carbono 2´ del azúcar carece del átomo de
oxigeno), bases nitrogenadas (adenina,
guanina, citosina o timina) y el grupo fosfato. El
DNA se encuentra en el núcleo de las células
eucarióticas y para su extracción es necesario:
un procedimiento mecánico para romper la
pared y membrana celular y así facilitar su La determinación de la concentración de DNA
extracción, es importante mencionar que el obtenido en mg/ mL se realizó por tres métodos
proceso debe ser llevado en condiciones en que diferentes:
se inhiban las enzimas degradativas de los Método 1. Se anexa Figura 1. Nomograma para
ácidos nucleicos (nucleasas), además de que el el cálculo de las concentraciones de ácidos
DNA obtenido debe ser purificado. nucleicos y proteínas.
Análisis de la técnica: Factor de dilución: 3
A 15 g de guayaba se le adiciono una solución Valor obtenido del nomograma para ácidos
de Tris/EDTA/NaCl, trisaminometano, que es un nucleicos: 0.016 mg / mL
regulador de pH, ácido
AISLAMIENTO DE DNA DE ORIGEN VEGETAL Y SU CARACTERIZACIÓN ESPECTROFOTOMÉTRICA
IDENTIFICACIÓN DE BASES PÚRICAS Y PIRIMÍDICAS POR CROMATOGRAFÍA EN CAPA FINA
𝑚𝑔
∴ Á𝑐𝑖𝑑𝑜𝑠 𝑛𝑢𝑐𝑙𝑒𝑖𝑐𝑜𝑠 = 3(0.016) = 0.048 ⁄𝑚𝐿
Método 2: Mediante la fórmula:
𝐴260 0.656 ∗ 3
[𝐷𝑁𝐴] = ∗ 𝐹𝐷 =
22500 22500
−5 𝑔 𝑚𝑔
= 8.74𝑥10 ⁄𝑚𝑙 𝑜 𝑏𝑖𝑒𝑛 0.0874 ⁄𝑚𝑙
Método 3: Mediante la relación:
50 𝜇𝑔 𝐷𝑁𝐴 𝑝𝑢𝑟𝑜 − 1 𝐴260
𝑥 − 𝐴260
∴ 𝑥 = 𝐴260 ∗ 50 𝜇𝑔 𝐷𝑁𝐴 𝑝𝑢𝑟𝑜 ∗ 𝐹𝐷
𝑥 = 0.656 ∗ 50 ∗ 3
𝜇𝑔 𝑚𝑔
= 98.4 ⁄𝑚𝑙 𝑜 𝑏𝑖𝑒𝑛 0.098 ⁄𝑚𝑙
Calculo de la pureza:
𝐴260 𝑛𝑚
𝑝𝑢𝑟𝑒𝑧𝑎 = ≈2
𝐴280 𝑛𝑚
0.656𝑛𝑚
𝑝𝑢𝑟𝑒𝑧𝑎 = = 0.9252
0.709𝑛𝑚
Discusión de resultados:
El espectro de absorción del DNA puro presenta
un máximo a una absorbancia de 260 nm que
corresponde a los ácidos nucleicos y un mínimo
en 230 nm correspondiente a enlaces
peptídicos, sin embargo, los datos de la
espectrofotometría indican un comportamiento
distinto al mencionado, pues la absorbancia a
230 nm y 280 nm son mayores a 260 nm, éste
resultado indica la presencia en mayor cantidad
de proteínas y enlaces peptídicos en
comparación al DNA
Potrebbero piacerti anche
- Norma API RP 500Documento23 pagineNorma API RP 500Alex Flores100% (11)
- Método What IfDocumento2 pagineMétodo What IfLi ClementeNessuna valutazione finora
- Seminario Practica 8Documento22 pagineSeminario Practica 8Alonso JimenezNessuna valutazione finora
- Fundamentos Físicos de Los Procesos Biológicos V3Documento446 pagineFundamentos Físicos de Los Procesos Biológicos V3lobocerna100% (10)
- Introduccion: ResultadosDocumento3 pagineIntroduccion: ResultadosLuis RojasNessuna valutazione finora
- Mecanica de Fluidos I Perdida de CargaDocumento42 pagineMecanica de Fluidos I Perdida de CargaJhonnyGonzalesJurado100% (1)
- Pract 3 Metodo de SangerDocumento3 paginePract 3 Metodo de SangerRay torresNessuna valutazione finora
- Calculo de Maxima DemandaDocumento9 pagineCalculo de Maxima DemandaAnonymous MMLzZjbyNessuna valutazione finora
- Reporte 8 QODocumento18 pagineReporte 8 QOrauljcNessuna valutazione finora
- Deshidrogena SuccínicaDocumento1 paginaDeshidrogena SuccínicaAngel BeltranNessuna valutazione finora
- Cuestionario 11Documento4 pagineCuestionario 11David Ramírez100% (1)
- Efecto de Concentración de Sustrato e Inhibición Enzimática.Documento2 pagineEfecto de Concentración de Sustrato e Inhibición Enzimática.H VandortNessuna valutazione finora
- Practica 2 Report eDocumento10 paginePractica 2 Report eEdson GonzalezNessuna valutazione finora
- Evaluación de Microorganismos Cultivables de AguaDocumento5 pagineEvaluación de Microorganismos Cultivables de AguaPanchoNessuna valutazione finora
- DnaDocumento2 pagineDnaSteele-GreyNessuna valutazione finora
- Seminario Sintesis A MicroescalaDocumento21 pagineSeminario Sintesis A MicroescalaFranco RoaNessuna valutazione finora
- Curva Tipo Azucares ReductoresDocumento2 pagineCurva Tipo Azucares ReductoresJeeniffer CookieeNessuna valutazione finora
- Efecto Del PHDocumento3 pagineEfecto Del PHClaw FollowilNessuna valutazione finora
- Practica 2 Ecologia MicrobianaDocumento5 paginePractica 2 Ecologia MicrobianaMiguel LunaNessuna valutazione finora
- PRACTICANITROGENODocumento9 paginePRACTICANITROGENOBrenda M.Nessuna valutazione finora
- Determinación de Aminoácidos Terminales Con Grupo Alfa Amino Libre.Documento1 paginaDeterminación de Aminoácidos Terminales Con Grupo Alfa Amino Libre.Gonzalo UribeNessuna valutazione finora
- Práctica Aislamiento de Dna PlásmidicoDocumento2 paginePráctica Aislamiento de Dna PlásmidicoLuis RojasNessuna valutazione finora
- Seminario Practica 5 Ácido FumaricoDocumento24 pagineSeminario Practica 5 Ácido FumaricojorgeNessuna valutazione finora
- Separación de FosfolípidosDocumento4 pagineSeparación de FosfolípidosAlain BleuNessuna valutazione finora
- Bitácora-Efecto de La Concentración de Enzima Sobre La Velocidad de Reacción-Efecto Del PH Sobre La Velocidad de ReacciónDocumento5 pagineBitácora-Efecto de La Concentración de Enzima Sobre La Velocidad de Reacción-Efecto Del PH Sobre La Velocidad de ReacciónDynarko RuizNessuna valutazione finora
- Separacionde Una Mezcla de Azul de MetilenoDocumento6 pagineSeparacionde Una Mezcla de Azul de MetilenogusNessuna valutazione finora
- Practica No. 3 "Análisis de La Calidad Del Agua II. Determinación Microbiológica"Documento4 paginePractica No. 3 "Análisis de La Calidad Del Agua II. Determinación Microbiológica"Brenda M.Nessuna valutazione finora
- Aislamiento y Caracterización Espectrofotométrica y Cromatografía en Capa Fina Del DnaDocumento2 pagineAislamiento y Caracterización Espectrofotométrica y Cromatografía en Capa Fina Del DnaGuillermo CasillasNessuna valutazione finora
- Seminario Equipo 3 Pract 2Documento38 pagineSeminario Equipo 3 Pract 2Gibran Meza CastrejonNessuna valutazione finora
- P12 ToxicidadDocumento4 pagineP12 ToxicidadJonathan R. EstradaNessuna valutazione finora
- Practica 1. SangerDocumento4 paginePractica 1. SangerApolinar Aguilar BautistaNessuna valutazione finora
- 3fm1 - Morales Vargas Citlali - Villada Victoria Vianney - Determinación de Aminoácidos Terminales Con Grupo Alfa Amino LibreDocumento4 pagine3fm1 - Morales Vargas Citlali - Villada Victoria Vianney - Determinación de Aminoácidos Terminales Con Grupo Alfa Amino LibreVianney VilladaNessuna valutazione finora
- Practica 6Documento2 paginePractica 6Vanesa Cx SlumberNessuna valutazione finora
- CUESTIONARIODocumento1 paginaCUESTIONARIOHector QNessuna valutazione finora
- Bioquimica Reacciones de Aminoacidos y ProteinasDocumento2 pagineBioquimica Reacciones de Aminoacidos y ProteinasEvelyn Jim 3295Nessuna valutazione finora
- Práctica 1 Transporte de Glucosa en LevadurasDocumento9 paginePráctica 1 Transporte de Glucosa en LevadurasReymundo SeguraNessuna valutazione finora
- Reporte Practica 8 FosfolípidosDocumento2 pagineReporte Practica 8 FosfolípidosAlex DVNessuna valutazione finora
- Seminario 6 Hidrolisis de Una ProteinaDocumento61 pagineSeminario 6 Hidrolisis de Una ProteinaLuisa RVNessuna valutazione finora
- DNA PlasmídicoDocumento2 pagineDNA PlasmídicoEduardo BeltranNessuna valutazione finora
- Caracterizacion de Glucogeno 2Documento3 pagineCaracterizacion de Glucogeno 2Jorge Luis OlivarNessuna valutazione finora
- Práctica 4. Adsorción II. Versión 2Documento4 paginePráctica 4. Adsorción II. Versión 2Gibran Meza CastrejonNessuna valutazione finora
- Practica No. 13Documento3 paginePractica No. 13fernando tvNessuna valutazione finora
- Practica Determinacion de Especies Alcalinas - Q. AnaliticaDocumento6 paginePractica Determinacion de Especies Alcalinas - Q. AnaliticaYoali Asiain LeconaNessuna valutazione finora
- Reacciones de Proteínas IDocumento3 pagineReacciones de Proteínas IEmriel96100% (1)
- Practica 5Documento18 paginePractica 5Gonzalo UribeNessuna valutazione finora
- SUCCÍNILDocumento3 pagineSUCCÍNILMarcos Jhoan OrtizNessuna valutazione finora
- Oxido-Reductasas 2Documento2 pagineOxido-Reductasas 2luis3felipe3lara3ortNessuna valutazione finora
- Separación de Fosfolípidos Por Cromatografía en Capa FinaDocumento4 pagineSeparación de Fosfolípidos Por Cromatografía en Capa FinaNahui morales lopezNessuna valutazione finora
- LDHDocumento2 pagineLDHENriqueNessuna valutazione finora
- Bioquimica Efecto PH y EnzDocumento3 pagineBioquimica Efecto PH y EnzGuerrillaScribdNessuna valutazione finora
- Practica 12 EcoDocumento4 paginePractica 12 EcoZuri ManriqueNessuna valutazione finora
- Práctica 3 Química BioorganicaDocumento1 paginaPráctica 3 Química BioorganicaSeccion 4 Maria Luisa Rosales VázquezNessuna valutazione finora
- Seminario Práctica 5 EcoDocumento29 pagineSeminario Práctica 5 EcoFernando VictoriaNessuna valutazione finora
- Práctica 1. Vías de AdministraciónDocumento17 paginePráctica 1. Vías de AdministraciónFernando Victoria100% (1)
- Pract 12 .ToxicidadDocumento3 paginePract 12 .ToxicidadmarthaNessuna valutazione finora
- Bitacora 4. Curva Tipo Azucares ReductoresDocumento3 pagineBitacora 4. Curva Tipo Azucares ReductoresJavier AnguianoNessuna valutazione finora
- Induccion de Mutantes en Escherichia Coli 1Documento84 pagineInduccion de Mutantes en Escherichia Coli 1Encb IpnNessuna valutazione finora
- ETS Métodos de Análisis 2021 ENCBDocumento12 pagineETS Métodos de Análisis 2021 ENCBCDL LNessuna valutazione finora
- Curva Tipo ColesterolDocumento2 pagineCurva Tipo ColesterolGuerrillaScribdNessuna valutazione finora
- Argentometria y Complexometria PracticasDocumento29 pagineArgentometria y Complexometria PracticasGustavo BarrazaNessuna valutazione finora
- Metodo de SangerDocumento3 pagineMetodo de SangerBlankita AguirreNessuna valutazione finora
- Separacion de FosfolipidosDocumento2 pagineSeparacion de Fosfolipidosironspidey10Nessuna valutazione finora
- Reporte 1 Fisiología CelularDocumento10 pagineReporte 1 Fisiología CelularMonserrat Blanquel GarcíaNessuna valutazione finora
- Practica 5 Potencial de Accion y Simulacion Por ComputadoraDocumento22 paginePractica 5 Potencial de Accion y Simulacion Por ComputadoraAndresDuranNessuna valutazione finora
- Cuantificación de Ácidos NucleicosDocumento9 pagineCuantificación de Ácidos NucleicosGustavoNessuna valutazione finora
- CALORDocumento2 pagineCALORLi ClementeNessuna valutazione finora
- ObjetivoDocumento2 pagineObjetivoLi ClementeNessuna valutazione finora
- Operaciones de Transferencia de MomentoDocumento1 paginaOperaciones de Transferencia de MomentoLi ClementeNessuna valutazione finora
- Equipos Más Utilizados en La IndustriaDocumento1 paginaEquipos Más Utilizados en La IndustriaLi ClementeNessuna valutazione finora
- Operaciones de Transferencia de MomentoDocumento1 paginaOperaciones de Transferencia de MomentoLi ClementeNessuna valutazione finora
- 31Documento14 pagine31Li ClementeNessuna valutazione finora
- ReligiónDocumento5 pagineReligiónLi ClementeNessuna valutazione finora
- Operaciones de Transferencia de MomentoDocumento1 paginaOperaciones de Transferencia de MomentoLi ClementeNessuna valutazione finora
- Tarea PescaditoDocumento3 pagineTarea PescaditoLi ClementeNessuna valutazione finora
- ElectrólisisDocumento3 pagineElectrólisisLi ClementeNessuna valutazione finora
- Diseño de Un Sistema de Adsorción en Carbón Activado para La Eliminación de Cromo Hexavalente en Disolución AcuosaDocumento1 paginaDiseño de Un Sistema de Adsorción en Carbón Activado para La Eliminación de Cromo Hexavalente en Disolución AcuosaLi ClementeNessuna valutazione finora
- Policloruro de Vinilo CloradoDocumento2 paginePolicloruro de Vinilo CloradoLi ClementeNessuna valutazione finora
- Diseño de Un Sistema de Adsorción en Carbón Activado para La Eliminación de Cromo Hexavalente en Disolución AcuosaDocumento1 paginaDiseño de Un Sistema de Adsorción en Carbón Activado para La Eliminación de Cromo Hexavalente en Disolución AcuosaLi ClementeNessuna valutazione finora
- Informe EmpresaDocumento2 pagineInforme EmpresaLi ClementeNessuna valutazione finora
- ElenaDocumento31 pagineElenaDavid Jaramillo KlinkertNessuna valutazione finora
- La AbsorciónDocumento2 pagineLa AbsorciónLi ClementeNessuna valutazione finora
- Taller de AdministracionDocumento1 paginaTaller de AdministracionLi ClementeNessuna valutazione finora
- Creatin ADocumento2 pagineCreatin ALi ClementeNessuna valutazione finora
- Proceso de Elaboración Del Hipoclorito de SodioDocumento2 pagineProceso de Elaboración Del Hipoclorito de SodioLi ClementeNessuna valutazione finora
- Creatin ADocumento2 pagineCreatin ALi ClementeNessuna valutazione finora
- KaibilDocumento2 pagineKaibilLi ClementeNessuna valutazione finora
- ElenaDocumento31 pagineElenaDavid Jaramillo KlinkertNessuna valutazione finora
- ObjetivoDocumento2 pagineObjetivoLi ClementeNessuna valutazione finora
- Guia de ValvulasDocumento14 pagineGuia de ValvulasAlejandro BriceñoNessuna valutazione finora
- Etil Engli ColDocumento1 paginaEtil Engli ColLi ClementeNessuna valutazione finora
- 5660 20758 1 SMDocumento8 pagine5660 20758 1 SMLi ClementeNessuna valutazione finora
- DestilacionDocumento1 paginaDestilacionFabrizio Ortiz PalzaNessuna valutazione finora
- Diet Il Engli ColDocumento1 paginaDiet Il Engli ColLi ClementeNessuna valutazione finora
- Acce So RiosDocumento2 pagineAcce So RiosLi ClementeNessuna valutazione finora
- 03 PROPIEDADES F SICAS DE LOS LENTES DE CONTACTO - PPSXDocumento14 pagine03 PROPIEDADES F SICAS DE LOS LENTES DE CONTACTO - PPSX1tv222tv22Nessuna valutazione finora
- Listado de SecuenciasDocumento25 pagineListado de Secuenciasjesus lopezNessuna valutazione finora
- FOSFATASASDocumento13 pagineFOSFATASASBîłłý Řôğěř100% (1)
- Práctica 6 - Relaciones de Masa en Los Procesos QuímicosDocumento4 paginePráctica 6 - Relaciones de Masa en Los Procesos QuímicosJoseMorilloNessuna valutazione finora
- D A V I D R A M o N D e L L I - CO-6°ABCF - Química - Actividad N°3-Primera EtapaDocumento11 pagineD A V I D R A M o N D e L L I - CO-6°ABCF - Química - Actividad N°3-Primera Etapad a v i d r a m o n d e l l iNessuna valutazione finora
- Recubrimientos MetalicosDocumento2 pagineRecubrimientos MetalicosEduardo RomoNessuna valutazione finora
- Cómo Funcionan Los Globos AerostaticosDocumento2 pagineCómo Funcionan Los Globos AerostaticosLupita Torres Guzman100% (1)
- 14 - PRIMER PRINCIPIO DE LA TERMODINÁMICA - Física en SegundosDocumento48 pagine14 - PRIMER PRINCIPIO DE LA TERMODINÁMICA - Física en SegundosManu FalcónNessuna valutazione finora
- Quimica Organica 5to AñoDocumento5 pagineQuimica Organica 5to AñoAnonymous kEC3kiy100% (2)
- Fisica Grado 11Documento15 pagineFisica Grado 11Juan Sebastian RodeloNessuna valutazione finora
- Introducción A Las Baterías de LitioDocumento27 pagineIntroducción A Las Baterías de LitioSORAYA MARTINEZ MAYGUANessuna valutazione finora
- PRESENTACION Dec 1584 VertimientoDocumento30 paginePRESENTACION Dec 1584 VertimientoErwin Guzman AurelaNessuna valutazione finora
- Práctica 4. Furfural Por Hidrolisis y Deshidratación de PentosasDocumento7 paginePráctica 4. Furfural Por Hidrolisis y Deshidratación de PentosasAlexia Berenice Gonzalez SanchezNessuna valutazione finora
- Presentación Electricidad PPDocumento12 paginePresentación Electricidad PPHéctor Plaza GonzálezNessuna valutazione finora
- Métodos ElectroquímicosDocumento7 pagineMétodos ElectroquímicosTaniaNessuna valutazione finora
- I. Introducción A La Refrigeración y Congelamiento Como Medio de Conservación de AlimentosDocumento13 pagineI. Introducción A La Refrigeración y Congelamiento Como Medio de Conservación de AlimentosBrayan Chavez OreNessuna valutazione finora
- SFMPDocumento4 pagineSFMPAdlaremse BlueNessuna valutazione finora
- RAMOS, OLIVER Actividad - 5 1NI122Documento12 pagineRAMOS, OLIVER Actividad - 5 1NI122Oliver RamosNessuna valutazione finora
- Parte ExperimentalDocumento3 pagineParte ExperimentalPamelaAltamiranoNessuna valutazione finora
- Sellado de FluidosDocumento93 pagineSellado de FluidosIrwin MartinNessuna valutazione finora
- Protocolo Toque y PasoDocumento1 paginaProtocolo Toque y Pasodayer100% (2)
- Contenido Del Primer ParcialDocumento76 pagineContenido Del Primer ParcialTermodinamica MoralesNessuna valutazione finora
- Lixiviacion Con Cloro y Thiosulfatos Final1)Documento16 pagineLixiviacion Con Cloro y Thiosulfatos Final1)Daniel CanalesNessuna valutazione finora
- In0706 Ingenieria de MaterialesDocumento6 pagineIn0706 Ingenieria de Materialesangeles condorNessuna valutazione finora
- CicloalcanosDocumento16 pagineCicloalcanosSebastian NamikazeNessuna valutazione finora
- Elementos GeologicosDocumento4 pagineElementos GeologicosMarcos CorderoNessuna valutazione finora